老面酵头又称“酸面团”、“老面”、“面肥”等[1-3],是将小麦粉或其他谷物粉与水混合后,自然发酵一定时间而成,在发酵食品中广泛应用[4]。老面酵头作为一种天然的发酵剂,是由乳酸菌和酵母菌等组成的复杂混合发酵体系[5],是一个微型的菌种资源库,通过微生物菌群间的酯化和糖化的协同效应生成多种风味物质,从而赋予老面馒头独特的风味和更高的营养价值[6-8]。此外,在发酵过程中还会生成蛋白酶、淀粉酶等酶类物质,对于面团的流变特性具有积极的改善作用,能够使馒头更加松软细腻,还可以延缓淀粉的老化,从而延长货架期[9]。目前,商业活性干酵母因其发酵时间短、便于控制,在馒头工业化生产中得到广泛的应用。但其菌种单一导致其风味远远不如老面馒头,由此可见,研究老面酵头中的微生物多样性至关重要。据现有报道,老面酵头中的乳酸菌的种类超过70种,其中常见的乳酸菌种有旧金山乳杆菌(Lactobacillus sanfranciscensis)、植物乳植杆菌(Lactiplantibacillus plantarum)、发酵乳杆菌(Lactobacillus fermentium)等;老面酵头中的酵母菌种类有25种之多,其中常见的酵母菌种有酿酒酵母(Saccharomyces cerevisiae)、矮小假丝酵母(Candida humilis)、光滑假丝酵母(Candida glabrata)等[10]。与国外对于老面酵头全面系统地研究相比,目前我国仍处于探索阶段,国内对于老面酵头的研究集中于某个地区或几个地区之间的菌群结构对比,关于活化对老面酵头微生物菌群多样性的影响研究较少。
自然界中的大多数微生物不能通过传统的平板培养法分离鉴定[10],而高通量测序技术可以直接从样品中提取脱氧核糖核酸(deoxyribonucleic acid,DNA)或核糖核酸(ribonucleic acid,RNA),通过聚合酶链式反应(polymerase chain reaction,PCR)扩增技术对细菌(16S rRNA)或真菌内部转录间隔(internal transcribed spacer,ITS)的可变区扩增后进行测序,从而获得样品的微生物菌群结构。高通量测序技术具有速度快、准确度高、测序量大、成本低等特点[11-12],目前,已在发酵食品、医学等领域得到了广泛应用[13-15],但在老面酵头中的菌群分析还鲜见报道。
本研究以老面酵头为研究对象,利用高通量测序技术,对不同活化时间和活化次数的老面酵头的微生物多样性进行分析,旨在为老面馒头建立品质稳定的生产工艺,实现高水平工业化生产提供参考。
1 材料与方法
1.1 材料与试剂
老面酵头:河北同福健康产业有限公司;小麦粉:五得利面粉集团有限公司;MagPure Soil DNA LQ Kit:广州美基生物科技有限公司;Qubit dsDNA Assay Kit:美国Life Technologies公司;Tks Gflex DNA Polymerase:宝生物工程(大连)有限公司。其他试剂均为国产分析纯或生化试剂。
1.2 仪器与设备
5418型台式高速离心机:德国Eppendorf公司;580BR 10905型PCR仪:美国Bio-rad公司;HE-120型电泳仪、Tanon 2500凝胶成像仪:上海天能科技有限公司;Aglient 2100 Bioanalyzer生物分析仪:美国Aglient公司;NanoDrop 2000核酸蛋白定量仪:赛默飞世尔科技有限公司。
1.3 方法
1.3.1 老面酵头样品的活化及取样
按照面粉∶水∶老面酵头=6∶3∶2(g∶g)的比例混合,置于26 ℃的条件下,每隔3 d进行一次活化培养,即每隔3 d取前一次发酵培养的老面酵头,在不额外加入其他微生物的情况下,反复地加入相同比例的新鲜面粉与水并混匀,共活化3次。原始老面酵头样品记为S0,第一次活化1 d老面酵头样品记为S1,第一次活化2 d老面酵头样品记为S2,第一次活化3 d老面酵头样品记为S3,第二次活化3 d老面酵头样品记为S4,第三次活化3 d老面酵头样品记为S5,在老面酵头的中心分散取样3次后置于-80 ℃冰箱中储存。
1.3.2 老面酵头微生物高通量测序分析
采用DNA抽提试剂盒对样本微生物的基因组DNA进行提取。利用NanoDrop 2000和琼脂糖凝胶电泳检测DNA的浓度和纯度,将提取的DNA保存于-20 ℃。以提取的基因组DNA为模板,使用带Barcode的特异引物和Tks Gflex DNA Polymerase高保真酶进行细菌16S rRNA基因和真菌ITS基因的PCR扩增。采用通用引物343F(5'-TACGGRAGGCAGCAG-3')和798R(5'-AGGGTATCTAATCCT-3')PCR扩增16S rRNA基因的V3~V4可变区,用于细菌菌群多样性分析[16]。采用通用引物ITS1F(5'-CTTGGTCATTTAGAGGAAGTAA-3')和ITS2R(5'-GCTGCGTTCTTCATCGATGC-3')扩增ITS基因的ITS1可变区[17],用于真菌菌群多样性分析。PCR扩增体系(30 μL):15 μL 2×Gflex PCR Buffer,1 μL上游引物(5 pmol/μL)和下游引物(5 pmol/μL),0.6 μL Tks Gflex DNA聚合酶,1 μL模板DNA,补加无菌水至总体积30 μL。PCR扩增程序:94 ℃预变性5 min;94 ℃变性30 s,56 ℃退火30 s,72 ℃延伸20 s,共26个循环;72 ℃再延伸5 min。PCR扩增产物使用琼脂糖凝胶电泳检测后,委托上海欧易生物技术有限公司使用Illumina NovaSeq 6000测序平台进行高通量测序。
1.3.3 序列处理与分析
通过UCHIME 2.4.2去除嵌合体序列,使用Vsearch 2.4.2[18]软件按照97%相似性标准对序列进行操作分类单元(operational taxonomic unit,OTUs)聚类分析。使用QIIME中的split_libraries 1.8.0[19]软件挑选出各个OTU的代表序列,然后利用RDP-classifier[20]软件以及Silva(version138)数据库和Unite数据库进行物种注释和分类学分析,置信阈值为70%。在欧易云分析平台(https://cloud.oebiotech.com/)进行多样性分析。
1.3.4 数据处理
采用SPSS Statistics 26.0软件对数据进行统计分析,采用Origin 2022软件绘制图表。
2 结果与分析
2.1 稀释曲线分析
稀释曲线不仅可以比较不同样本中物种的丰富度,还能够反映测序深度是否合理。若曲线平缓则说明测序数据量合理,反之则表明测序数据不够合理,测序深度不够[10]。因此,从样本中随机抽取一定数量的序列,统计其所对应的物种数目(Sobs指数与Shannon指数),并以抽取的序列数与物种数绘制稀释曲线,结果见图1。由图1可知,随着序列数的增加,老面酵头样品微生物Sobs指数与Shannon指数稀释曲线都呈急剧升高后逐渐趋于平缓的趋势,表明样品测序深度合理,所取样本量能够覆盖绝大多数的菌种信息。
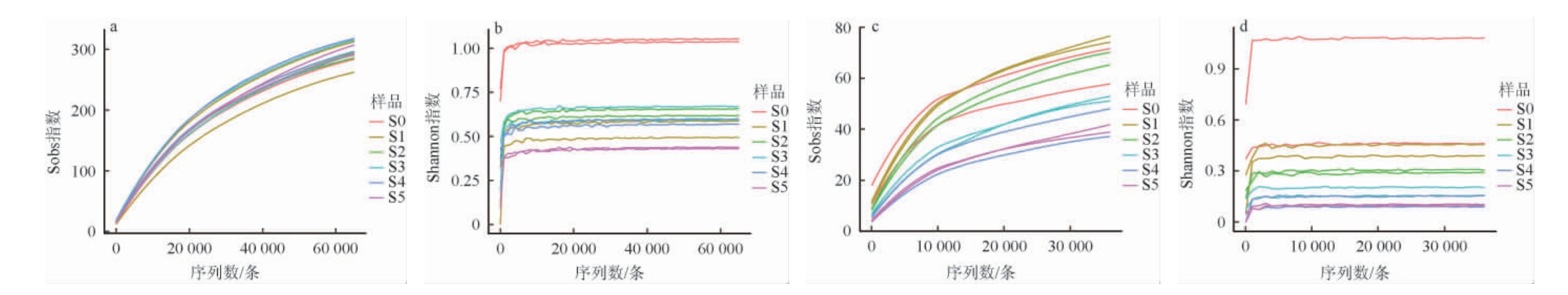
图1 老面酵头样品细菌及真菌的Sobs指数(a、c)及Shannon指数(b、d)稀释曲线
Fig.1 Sobs index (a, c) and Shannon index (b, d) rarefaction curves of bacteria and fungi in sourdough samples
2.2 微生物菌群多样性分析
Alpha多样性分析可以反映样品中物种的丰富度和均匀度,Alpha多样性指数主要包括:Chao1、Ace、Shannon、Simpson、Coverage和Sobs指数等[21]。其中,Chao1和Ace指数估算样品中所含的OTU数目,反映物种的丰富度;Shannon和Simpson指数反映菌群的多样性;Sobs指数反映实际观测到的OTU数目,Coverage指数代表样品测序文库的覆盖率,可以用来评估测序结果能否代表样本中菌群结构的实际情况。不同活化时间及活化次数老面酵头样品微生物的Alpha多样性指数见表1。
表1 老面酵头样品中微生物菌群的Alpha多样性分析结果
Table 1 Results of Alpha diversity analysis of microbial flora in sourdough samples

注:同列间不同小写字母表示差异显著(P<0.05)。
微生物 样品名称 Sobs指数 Shannon指数 Simpson指数 Ace指数 Chao1指数 Coverage指数/%细菌真菌S0 S1 S2 S3 S4 S5 S0 S1 S2 S3 S4 S5 288.10±5.37a 287.30±34.22a 291.85±6.72a 304.60±17.68a 307.60±16.12a 300.05±12.09a 64.40±10.75ab 75.45±2.05a 68.50±2.12a 50.85±2.19bc 44.25±8.41c 41.00±2.12c 1.05±0.01a 0.54±0.07c 0.64±0.03b 0.63±0.05bc 0.58±0.02bc 0.43±0.01d 0.77±0.44a 0.42±0.05ab 0.30±0.01b 0.18±0.03b 0.12±0.05b 0.10±0.01b 0.31±0.01a 0.11±0.01d 0.13±0.01b 0.13±0.01bc 0.11±0.00cd 0.08±0.00e 0.17±0.11a 0.08±0.01ab 0.06±0.00b 0.03±0.01b 0.02±0.01b 0.02±0.00b 412.12±20.00a 414.89±14.37a 414.54±40.38a 434.85±6.03a 417.61±6.04a 450.64±63.73a 84.33±24.03abc 91.09±0.57a 90.15±1.42ab 73.90±0.25abc 61.96±9.62bc 56.70±7.62c 397.29±28.92a 387.97±22.93a 420.71±50.23a 427.94±20.34a 406.72±5.49a 422.70±51.84a 77.71±18.80ab 90.62±5.83a 86.72±0.10ab 67.30±1.75ab 64.36±12.78ab 60.65±7.58b 99.83±0.00a 99.83±0.00a 99.82±0.00a 99.82±0.00a 99.83±0.00a 99.81±0.00a 99.96±0.00a 99.95±0.00a 99.94±0.00a 99.96±0.00a 99.96±0.00a 99.96±0.00a
由表1可知,所有老面酵头样品微生物菌群的Coverage指数均>99.81%,说明本次测序结果能够真实地反映样品中微生物菌群的实际情况。针对细菌,各样品细菌菌群的Sobs指数无显著性差异(P>0.05),说明各样品实际观测到的OTU数目无显著性差异;Ace指数和Chao1指数也无显著性差异(P>0.05),说明各样品的细菌菌群丰富度无显著性差异,即活化时间及次数对老面酵头中细菌菌群的丰富度无显著性影响。Shannon指数越大,表明样品中的群落多样性越高[10]。原始老面酵头样品(S0)细菌菌群的Shannon指数最大为1.05,第三次活化3 d的老面酵头样品(S5)细菌菌的Shannon指数最小为0.43,说明活化降低了老面酵头细菌菌群多样性。Simpson指数是指随机抽取两个个体属于不同物种的概率,其数值越高,表明群落多样性越高[10]。随着活化时间的延长,Simpson指数呈现先下降后上升的趋势,原始老面酵头样品(S0)的Simpson指数最高为0.31;随着活化次数的增加,Simpson指数呈现下降的趋势,说明活化对老面酵头样品细菌菌群的多样性有不利的影响。
针对真菌,随着老面酵头的活化,Sobs指数呈先上升后下降的趋势,第一次活化1 d老面酵头样品(S1)的Sobs指数最高为75.45,第三次活化3 d老面酵头样品(S5)的Sobs指数最低为41.00。随着活化时间的延长,Ace指数和Chao1指数均呈先上升后下降的趋势,第一次活化1 d老面酵头样品(S1)的Ace指数和Chao1指数最高,分别为91.09和90.62,说明在第一次活化1 d时老面酵头样品的真菌菌群丰富度最高,可能是因为新鲜面粉的添加给部分菌种带来了有利的生存条件;在活化3 d时,Ace指数和Chao1指数下降,真菌菌群丰富度降低,可能是于微生物相互竞争营养物质,使部分微生物的生长受到了抑制。随着活化次数的增多,Ace指数和Chao1指数均呈下降的趋势,说明活化对于老面酵头真菌菌群丰富度存在不利的影响。随着老面酵头的活化,Shannon指数和Simpson指数均呈现下降的趋势,原始老面酵头样品(S0)的Shannon指数和Simpson指数指数最大,分别为0.77和0.17,第三次活化3 d,老面酵头样品(S5)的Shannon指数和Simpson指数指数最小,分别为0.10和0.02,说明活化降低了老面酵头真菌菌群的多样性。
综上,随着活化时间的增加,老面酵头的细菌菌群丰富度无显著变化(P>0.05),多样性呈现先降低后升高的趋势;真菌菌群丰富度呈先升高后下降的趋势,多样性呈下降趋势。随着活化次数的增加,老面酵头的细菌菌群丰富度无显著变化(P>0.05),真菌群菌群丰富度及细菌和真菌菌群多样性均呈下降趋势,说明活化对于老面酵头的菌群多样性具有不利的影响。
2.3 微生物菌群结构分析
2.3.1 细菌菌群组成分析
不同活化时间及活化次数老面酵头样品的细菌菌群组成见图2。由图2a可知,在门水平上,从老面酵头样品中共检测出16个细菌门,包括3个优势细菌门,分别为厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidota)和变形菌门(Proteobacteria),这与陶康等[22-24]的研究结果基本吻合。厚壁菌门在S0、S1、S2、S3、S4和S5样品中的相对丰度分别为99.09%、99.17%、99.10%、99.05%、98.79%和98.68%,是老面酵头样品中的绝对优势细菌门;拟杆菌门在S0、S1、S2、S3、S4和S5样品中的相对丰度分别为0.59%、0.56%、0.55%、0.56%、0.55%和0.46%;变形菌门在S0、S1、S2、S3、S4和S5样品中的相对丰度分别为0.14%、0.12%、0.13%、0.13%、0.21%和0.27%。
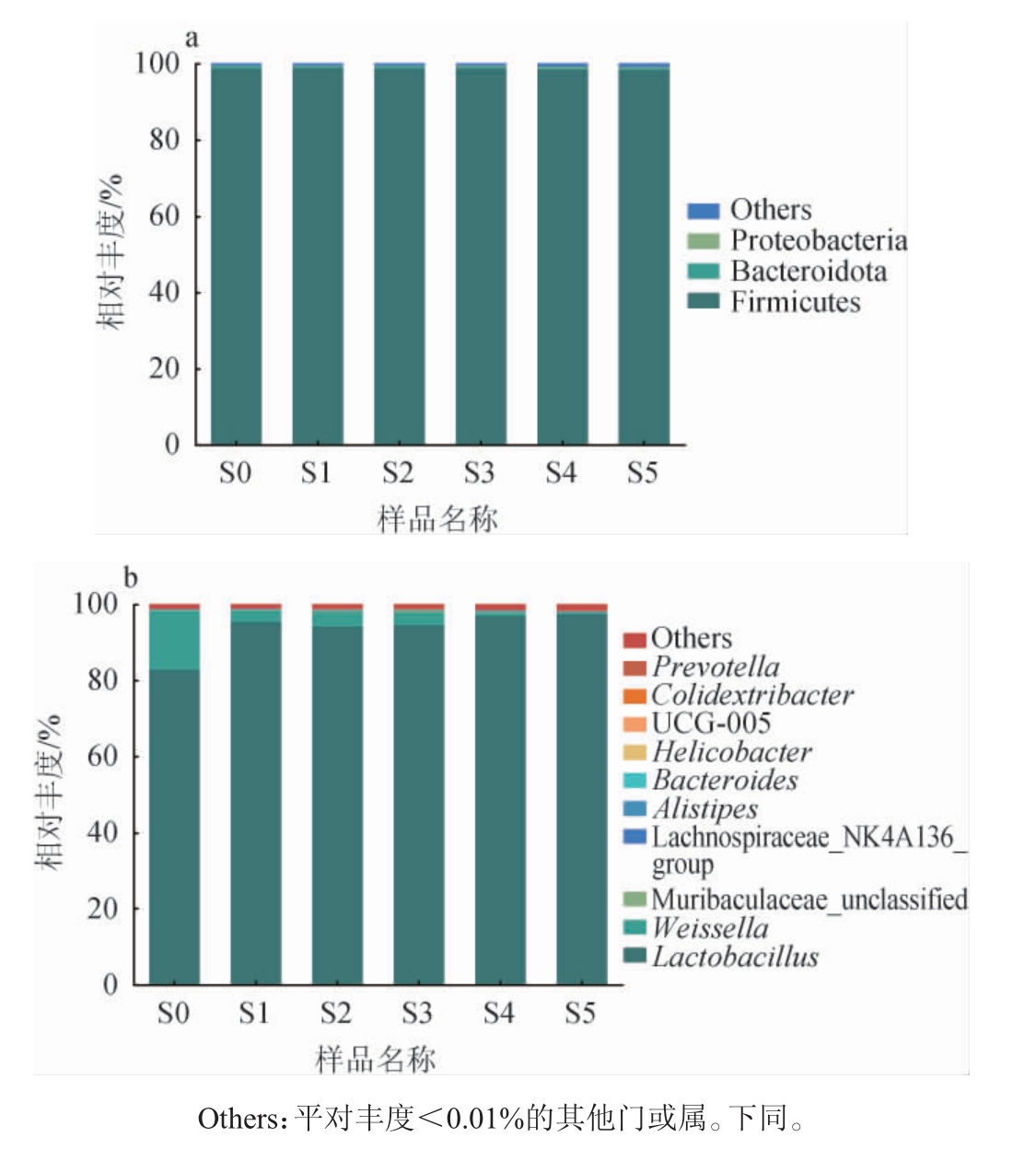
图2 基于门(a)和属(b)水平老面酵头样品中的细菌菌群结构
Fig.2 Structure of bacterial flora in sourdough samples based on phylum (a) and genus (b) levels
由图2b可知,在属水平上,从老面酵头样品中共检测出195个细菌属,包括2个优势细菌属,分别为乳杆菌属(Lactobacillus)和魏斯氏菌属(Weissella)。其中,乳杆菌属(Lactobacillus)相对丰度最高,为绝对优势细菌属。不同样品优势细菌属的相对丰度差异明显。在第一次活化过程中,Lactobacillus的相对丰度呈先上升后略下降的趋势,随着活化时间的延长,其相对丰度迅速增加。在第一次活化1 d时,相对丰度最高,为95.72%,可能是新物料的添加有利于Lactobacillus的生长;在第一次活化2 d和3 d时,Lactobacillus的相对丰度略有下降,基本稳定在94%以上。第二、三次活化时,Lactobacillus的相对丰度比第一次活化略有上升,稳定在97%以上。与其他研究的结果一致,乳杆菌属是老面酵头中的主要优势菌属[25-27],这可能与乳杆菌属具有独特的基因组特征有关,还可能因为其细胞内的碳水化合物活性酶存在特异性,使其对碳水化合物高度适应并能够加以代谢[28],从而增强了它的竞争力[29],成为老面酵头中的主要优势菌属。乳杆菌属普遍存在于传统发酵剂中,能够很好的利用面粉中的主要糖类物质,对老面馒头的口感、风味以及营养价值具有十分重要的影响[30-32]。在第一次活化过程中,魏斯氏菌属(Weissella)总体呈现下降的趋势,随着活化时间的延长,其相对丰度下降。在第一次活化1 d时Weissella的相对丰度迅速下降,由15.36%下降至2.87%;在第一次活化2 d和3 d时,Weissella的相对丰度略有上升,稳定在3%~4%。第二、三次活化时,Weissella的相对丰度呈下降趋势,第三次活化3 d时,相对丰度最低,为0.15%。老面酵头的水分含量较低,且长期存放在室温条件下,魏斯氏菌属(Weissella)具有产胞外多糖的能力,使其能够保持较好的生理活性[33]。总体而言,不同活化时间及活化次数老面酵头样品的优势细菌属相对丰度差异显著,表明活化时间及活化次数在一定程度上均能够影响优势细菌属的结构分布和演替规律。
2.3.2 真菌菌群组成分析
不同活化时间及活化次数的老面酵头样品的真菌菌群组成见图3。由图3a可知,在门水平上,从老面酵头样品中共检测出4个真菌门,包括3个优势真菌门,分别为子囊菌门(Ascomycota)、担子菌门(Basidiomycota)和球囊菌门(Glomeromycota)。子囊菌门在S0、S1、S2、S3、S4和S5样品中的相对丰度分别为99.41%、99.88%、99.92%、99.96%、99.98%和99.99%,是老面酵头样品中的绝对优势真菌门,与刘明伟[23]的研究结果基本一致。随着活化时间和活化次数的增加,子囊菌门在样品中的相对丰度逐渐增加。担子菌门在S0、S1、S2、S3、S4和S5样品中的相对丰度分别为0.24%、0.05%、0.05%、0.02%、0.01%和0.01%。与子囊菌门相反,担子菌门在样品中的相对丰度随着活化时间和活化次数的增加而减少。球囊菌门在S0、S1、S2、S3、S4和S5样品中的相对丰度分别为0.029%、0.011%、0.004%、0.004%、0.001%和0.002%。
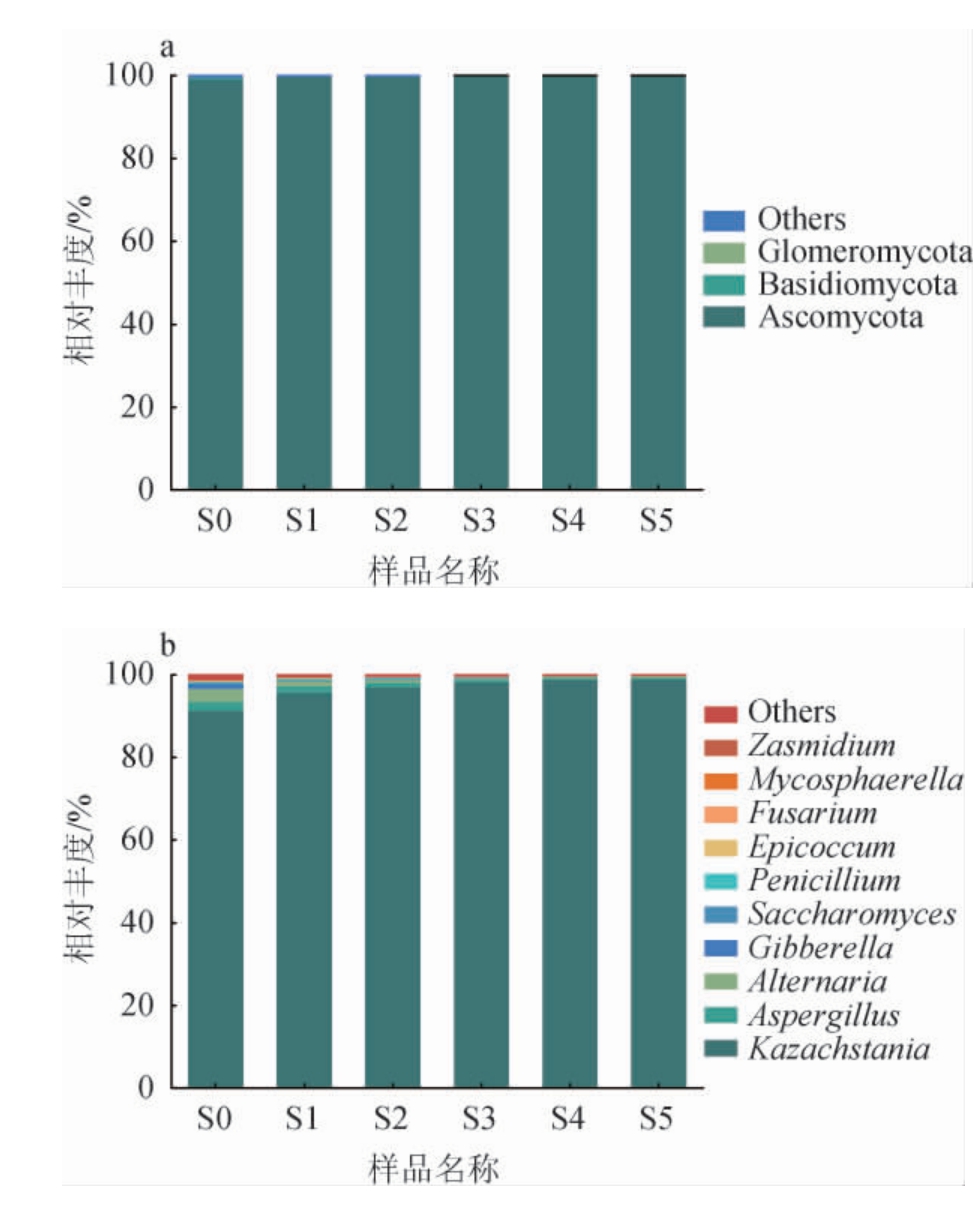
图3 基于门(a)和属(b)水平老面酵头样品中的真菌菌群结构
Fig.3 Structure of fungal flora in sourdough samples based on phylum (a) and genus (b) levels
由图3b可知,在属水平上,从老面酵头样品中共检测出46个真菌属,包括3个优势真菌属,分别为哈萨克斯坦酵母属(Kazachstania)、交链孢霉属(Alternaria)和曲霉属(Aspergillus)。其中,哈萨克斯坦酵母属(Kazachstania)相对丰度最高,为绝对优势真菌属。不同样品优势属的相对丰度差异明显。在活化过程中,Kazachstania的相对丰度呈上升趋势,由91.48%上升至99.17%。Kazachstania无法利用面粉中的麦芽糖,与乳杆菌属不存在对相同碳水化合物的竞争,所以在老酵面团的整个发酵期间能够与乳杆菌属具有良好的共生关系[34]。交链孢霉属(Alternaria)、曲霉属(Aspergillus)、赤霉属(Gibberella)、酵母属(Saccharomyces)、附球菌属(Epicoccum)、镰刀菌属(Fusarium)与Kazachstania呈相反的趋势。曲霉属能够使老酵面团产生多种酶类物质,可以将老面酵头中的氨基酸和单糖提供给酵母菌和乳酸菌进行代谢后利用[33]。青霉属(Penicillium)呈先上升后下降的趋势,在第一次活化1 d时,Penicillium的相对丰度最高,为0.32%,最终在第三次活化3 d时,相对丰度最低,为0.07%。
2.4 基于属水平老面酵头样品聚类分析
为更加直观地分析同一分类水平下不同样本之间的异同,基于属水平,分别选取平均相对丰度排名前30的细菌属和真菌属对老面酵头样品进行聚类分析,绘制热图,结果见图4。

图4 基于细菌(a)和真菌(b)属水平老面酵头样品聚类分析热图
Fig.4 Heatmap for cluster analysis of sourdough samples based on bacterial (a) and fungal (b) genus levels
由图4a可知,不同活化时间及活化次数的老面酵头样品中相对丰度较高的细菌属差异较大,Weissella、普雷沃氏菌属(Prevotella)、梭菌科(Clostridia)_UCG-014主要存在于S0样品中;Muribaculum、链球菌属(Streptococcus)、瘤胃球菌属(Ruminococcus)主要存在于S1样品中;拟杆菌属(Bacteroides)、毛螺菌科(Lachnospiraceae)_UCG-001主要存在于S2样品中;拟普雷沃氏菌属(Alloprevotella)、幽门螺旋杆菌属(Helicobacter)、颤杆菌属(Oscillibacter)主要存在于S3样品中;鞘氨醇单胞菌属(Sphingomonas)、Lachnoclostridium、杜博氏菌属(Dubosiella)主要存在于S4样品中;葡萄球菌属(Staphylococcus)、不动杆菌属(Acinetobacter)主要存在于S5样品中。
由图4b可知,S0样品具有较其他老面独特的菌群组成,Alternaria、Aspergillus、Gibberella、Saccharomyces、Epic occum、Fusarium、Zasmidium、球腔菌属(Mycosphaerella)等主要存在于S0样品中;Penicillium、Hamigera、葡柄霉属(Stemphylium)主要存在于S1样品中;枝顶孢属(Acremonium)主要存在于S2样品中;Xerochrysium主要存在于S3样品中;S4和S5样品的高丰度菌属相似,均为Kazachstania。
3 结论
利用高通量测序技术对不同活化时间和活化次数老面酵头样品的细菌和真菌菌群多样性进行分析,结果发现,随着活化时间的增加,老面酵头的细菌菌群丰富度无显著变化(P>0.05),多样性呈先降低后升高的趋势,真菌菌群丰富度呈先升高后下降的趋势,多样性呈下降趋势;随着活化次数的增加,老面酵头的细菌菌群丰富度无显著变化(P>0.05),真菌群菌群丰富度及细菌和真菌菌群多样性均呈下降趋势,说明活化对于老面酵头的菌群多样性具有不利的影响,老面酵头内部菌群结构复杂,受环境等影响较大。不同活化时间和活化次数老面酵头样品中相对丰度较高的微生物属存在一定差异;从老面酵头样品中共检测到16个细菌门、195个细菌属及4个真菌门、46个真菌属,优势细菌门分别为厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidota)和变形菌门(Proteobacteria);优势细菌属分别为乳杆菌属(Lactobacillus)和魏斯氏菌属(Weissella);优势真菌门分别为子囊菌门(Ascomycota)、担子菌门(Basidiomycota)和球囊菌门(Glomeromycota);优势真菌属分别为哈萨克斯坦酵母属(Kazachstania)、交链孢霉属(Alternaria)和曲霉属(Aspergillus)。本研究确定了老面酵头的优势菌种组成,可以为老面馒头工业建立品质稳定的生产工艺,实现高水平工业化生产提供参考,为后续构建老面酵头微生物菌群结构与馒头风味和品质之间的相关性奠定了基础。但对于构建老面酵头微生物菌群结构与馒头风味以及品质之间的相关性,老面酵头中优势菌种的分离纯化,以及老面酵头菌群结构的改善,仍需要进一步研究。
[1]李晓敏,韩伟,黎琪,等.传统酸面团菌群结构及其酵制馒头风味物质分析[J].食品科学,2021,42(10):162-170.
[2]马学利,李绍嵩,乔通通,等.传统馒头发酵剂菌群结构与菌种开发研究进展[J].粮食与油脂,2023,36(1):8-11.
[3]张国华,何国庆.我国传统馒头发酵剂的研究现状[J].中国食品学报,2012,12(11):115-120.
[4]CHAVAN R S,CHAVAN S R.Sourdough technology-a traditional way for wholesome foods:a review[J].Compr Rev Food Sci Safety,2011,10:169-182.
[5]ZHANG J C, LIU W J, SUN Z H, et al.Diversity of lactic acid bacteria and yeasts in traditional sourdoughs collected from western region in Inner Mongolia of China[J].Food Control,2011,22(5):767-774.
[6]杨敬雨,刘长虹.中国传统酵子的工业化[J].食品研究与开发,2007,28(2):164-166.
[7]PÉTEL C,ONNO B,PROST C,Sourdough volatile compounds and their contribution to bread: A review[J].Trends Food Sci Tech, 2017, 59:105-123.
[8]TANG X J, LIU R S, HUANG W N, et al.Impact of in situ formed exopolysaccharides on dough performance and quality of Chinese steamed bread[J].LWT-Food Sci Technol,2018,96:519-525.
[9]LIU T J, LI Y, CHEN J C, et al.Prevalence and diversity of lactic acid bacteria in Chinese traditional sourdough revealed by culture dependent and pyrosequencing approaches[J].LWT-Food Sci Technol,2016,68:91-97.
[10]邢小龙.河南地区老酵面团菌群结构及优势菌种复配研究[D].郑州:河南农业大学,2020.
[11]施慧琳,苏燕,许丽,等.高通量测序行业现状与发展趋势分析[J].生物产业技术,2018,10(3):6-12.
[12]TAGO K,ITOH H,KIKUCHI Y,et al.A fine-scale phylogenetic analysis of free-living Burkholderia species in sugarcane field soil[J].Microbes Environ,2014,29(4):434-437.
[13]YE J,JOSEPH S D,JI M,et al.Chemolithotrophic processes in the bacterial communities on the surface of mineral-enriched biochars[J].ISME J,2017,11(5):1087-1101.
[14]刘飞翔,董其惠,吴蓉,等.不同国家和地区传统发酵食品及其发酵微生物研究进展[J].食品科学,2020,41(21):338-350.
[15]杨光萍.高通量测序技术在食品微生物检测中的应用[J].食品安全导刊,2022,16(30):182-185.
[16]NOSSA C W,OBERDORF W E,YANG L,et al.Design of 16S rRNA gene primers for 454 pyrosequencing of the human foregut microbiome[J].World J Gastroenterol,2010,16(33):4135-4144.
[17]MUKHERJEE P K,CHANDRA J,RETUERTO M,et al.Oral mycobiome analysis of HIV-infected patients:identification of Pichia as an antagonist of opportunistic fungi[J].PLoS Pathog,2014,10(3):e1003996.
[18]CAPORASO J G, KUCZYN J, STOMBAUGH J, et al.QIIME allows analysis of high-throughput community sequencing data[J].Nat Methods,2010,7(5):335-341.
[19]ROGNES T, FLOURI T, NICHOLS B, et al.VSEARCH: a versatile open source tool for metagenomics[J].Peer J,2016,4:e2584.
[20]WANG Q,GARRITY G M,TIEDJE J M,et al.Naive Bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy[J].Appl Environ Microbiol,2007,73(16):5261-5267.
[21]李艳婷,郭尚,郭霄飞,等.清香型白酒发酵过程中微生物群落结构变化及与理化性质的相关性[J].中国食品学报,2023,23(5):330-340.
[22]陶康,吴凌伟,金晓芳,等.基于高通量基因测序分析腐乳微生物多样性[J].食品科学,2021,42(8):143-149.
[23]刘明伟.基于高通量基因测序分析酸面团微生物及复合发酵馒头研制[D].南昌:南昌大学,2022.
[24]LIU X J, ZHOU M, CHEN J X, et al.Bacterial diversity in traditional sourdough from different regions in China[J].LWT-Food Sci Technol,2018,96:251-259.
[25]李梁,薛蓓,罗章,等.基于高通量测序分析冻干老面与新鲜老面细菌群落结构[J].中国食品添加剂,2018,29(10):126-131.
[26]郭淑文,石晶红.基于高通量测序分析内蒙古地区传统酸面团中细菌菌群的多样性[J].中国酿造,2022,41(12):86-90.
[27]白雪.辽东地区酸汤子面团微生物多样性分析及其对面包品质的影响[D].沈阳:沈阳师范大学,2020.
[28]SWANSON K S, GIBSON G R, HUTKINS R, et al.The International Scientific Association for Probiotics and Prebiotics (ISAPP) consensus statement on the definition and scope of synbiotics[J].Nat Rev Gastroen Hepat,2020,17(11):687-701.
[29]VUYST L D, HARTH H, KERREBROECK S V, et al.Yeast diversity of sourdoughs and associated metabolic properties and functionalities[J].Int J Food Microbiol,2016,239:26-34.
[30]刘同杰.传统酸面团中微生物多样性及其风味物质代谢研究[D].杭州:浙江大学,2018.
[31]高静,刘敏,吴丹丹,等.基于高通量测序分析老面中菌群多样性及其对馒头品质的影响[J].中国粮油学报,2019,34(7):13-19.
[32]李佳慧.老酵馒头微生物种间关系及代谢产物的研究[D].天津:天津科技大学,2017.
[33]闫博文.老面酵头微生物菌群多样性差异分析及其对馒头风味特性的影响[D].无锡:江南大学,2019.
[34]YAN B, SADIQ F A, CAI Y J, et al.Microbial diversity in traditional type I sourdough and jiaozi and its influence on volatiles in Chinese steamed bread[J].LWT-Food Sci Technol,2019,101:764-773.