高粱(Sorghum bicolor L.)为禾本科(Poaceae)高粱属(Sorghum sp.)植物,是我国广泛种植的五谷类作物,也是酿酒业的重要原料[1]。酒的品质与高粱质量密不可分,如高粱品种红缨子是茅台镇特有的有机绿色无公害糯高粱,其淀粉含量高、组织紧密、纤维层粗厚,出酒效率高、酒质好,是茅台酒传统酿造工艺的最佳优选原料[2]。优质高粱的生长与当地气候环境和土壤条件息息相关,土壤微生物不仅对土壤有机质合成和营养物质转化具有重要作用,也是预测土壤质量变化的重要指标之一[3]。
土壤是微生物生活的大本营,是微生物资源多样性的宝库[4]。根与土壤直接“对话”,并连同根际(距离根约1 mm土壤范围)从土壤中招募、定殖对植物有益的微生物区系[5]。土壤中的微生物富集于根际并进入根内,进而形成一定的微生物区系,并且这种微生物区系的形成受多种因素影响。EDWARDS J等[6]对水稻根系的微生物组进行研究,发现土壤中微生物进入根内需要经过根际和根表面两道“关卡”的筛选,其中根表面的选择压力要远大于根际。同时,从根际到根内所定殖的微生物受土壤类型的影响最大,其次是水稻生长周期和品种。WILLIAM A W等[7]使用大规模种植的玉米为材料并基于大样本分析发现,定殖于根际的微生物中存在一些与品系高度关联的核心微生物,并且对于植物而言是可遗传的。XU L等[8]在大田环境中研究干旱对高粱根系微生物组建立的影响机制时,发现在植株不同生长阶段,干旱会延迟高粱生长前期时根内微生物组的形成,并影响相关代谢活动。ZHAO L等[9]研究3种不同酒用高粱根微域(根际、根面和根内)细菌群落多样性及组装过程机制,发现细菌沿土壤-根际-根面-根内空间路径的组装机制以同质性选择为主,进入过程受到的扩散限制逐渐增强,菌群之间的网络连接关系逐渐减弱,且根面是根组织对土壤细菌群进行选择的关键屏障。
ZHAOZZ等[10-13]针对作物土壤、酒醅和油污土壤中的各类优势菌群和稀有菌群进行研究,发现总群落(entire community,EC)中的这些亚群落在组装机制和多样性形成中与总群落相比有一定差异,且对总群落的最终形成产生关键作用,进而表明总群落内部不同亚群落有其特有的形成机制,并最终决定总群落的形成。因此,可以推测,细菌从土壤进入根组织的过程中,总群落的形成应该是其各类亚群落共同作用的结果,而其中的亚群落形成、以及与环境因素(如土壤理化因素)间的响应关系与总群落相比有一定的区别。厘清各亚群落的形成过程将对红缨子高粱根系微生物的构建过程有一个更全面的认识。基于上述假设,本研究利用Illumina MiSeq高通量测序技术获得土壤、根际、根面和根内4种根系组分的总群落(EC),并从中分离核心群落(core community,CC)和特有群落(unique community,UC)数据,分别从多样性特征、土壤养分因子响应关系,以及群落结构等几方面,比较分析核心群落和特有群落与总群落的区别,以及它们对总群落形成的角色和作用,为科学、系统的理解红缨子高粱根系微生物形成特征提供参考。
1 材料与方法
1.1 材料与试剂
1.1.1 材料
红缨子高粱于当地高粱种植季4月底(2022年)开始种植,种植地点位于贵州省仁怀市鲁班镇茅台学院实验田,年平均气温16.3 ℃,年平均降雨量在800~1 000 mm。种植区土壤为紫红壤,面积约363 m2(11 m×33 m)。
1.1.2 化学试剂
Fast脱氧核糖核酸(deoxyribonucleic acid,DNA)R SPIN Kit:美国MP Biochemicals公司;ALPHA-SEQ Advanced Water DNA Kit:美国Kapa Biosystems公司;Minkagene plant DNA Kit:日本Minkabio公司;琼脂糖:西班牙Biowest公司;GeneJET Gel Extraction Kit:美国Thermo Fisher Scientific公司;磷酸盐缓冲液(phosphate buffer saline,PBS):北京索莱宝公司。其他试剂均为国产分析纯。
1.2 设备与仪器
Sigma 3K-15冷冻离心机:德国Sigma公司;DS-031S超声振荡仪:广东固特超声股份有限公司;NanoDrop2000超微量分光光度计:美国Thermo Fisher Scientific公司;ABI GeneAmpe 9700型聚合酶链式反应(polymerase chain reaction,PCR)仪:美国ABI公司;Illumina Miseq测序仪:美国Illumina公司。
1.3 方法
1.3.1 根系及土壤样品采集及前处理
待高粱生长到苗期(生长期约15 d)开始取根系样品,每10~15个植株根系合并为一个样品,依此共取5个根系样品,并根据EDWARDS J等[6,14]方法收集根际土、根面土和根组织样品。同时,从距种植区域约5~10 m的非种植区(无作物种植)的四角和中心各取一个土壤样品,作为种植本体土(bulk soil)。
1.3.2 土壤养分指标的测定
参考JIAO S等[15-16]方法测定根际土和本体土的养分指标,包括pH值、速效钾(available potassium,effK)、速效磷(available phosphorus,effP)、速效氮(available nitrogen,effN)、硝态氮(nitrate-nitrogen,noN)、铵态氮(ammoniumnitrogen,nhN)、总有机碳(total organic carbon,TOC),以及可交换硼(B)、锰(Mn)、铁(Fe)、锌(Zn)、铜(Cu)、钙(Ca)、镁(Mg)、钠(Na)的含量。
1.3.3 微生物总DNA提取
利用Illumina Miseq高通量测序技术对细菌群落的16S rRNA基因进行测序分析[9]。利用Fast DNAR SPIN Kit、ALPHA-SEQ Advanced Water DNA Kit和Minkagene plant DNA kit分别从本体土和根际土、根面土、根组织中提取微生物总DNA,以其为模板,使用引物对319F(5'-ACTCCTACGGGAGGCAGCAG-3')和806R(5'-GGACTACHVGGGTWTCTAAT-3')[17]PCR扩增16S rRNA基因的V3-V4区域基因序列。所有样品均进行3次PCR扩增,所有步骤均包含无模板对照。3次PCR扩增产物混合后,采用2%琼脂糖凝胶进行电泳分析。具有明亮条带的PCR扩增产物按等比例混合,然后用GeneJET Gel Extraction Kit纯化。纯化后的PCR扩增产物在微科盟生物科技有限公司的Illumina MiSeq 300平台上进行高通量测序,以获得本体土、根际、根面和根内4种根系组分细菌群落的扩增序列异变体(amplicon sequence variants,ASVs)数据信息。
1.3.4 序列处理及统计分析
使用Fastp(v0.14.1)软件包对序列进行拼接和去冗余,采用Fastqc软件包控制序列质量[18]。利用Qiime2-2021.8的DADA2算法生成ASVs,并对ASVs序列去噪[19-21]。ASVs片段与Silva数据库比对(silva138)生成带分类注释的ASVs丰度表[21]。使用Qiime2的filter-seqs和filter-table函数去除与"Chloroplast"和"mitochondria"匹配的富集数据[22],然后使用Phylogeny(align-to-tree-mafft-fasttree)函数构建ASVs代表序列的系统发育树[23-24]。
所有统计分析及作图均使用R软件(v4.2.2)[25]各类程序包完成。参考赵亮等[26]的方法,细菌群落的α-多样性采用系统进化多样性的平均成对谱系距离(mean pairwise phylogenetic distance,MPD)指数[27]和香农(Shannon)指数来衡量;β-多样性由群落间Bray-Curtis距离衡量。参考FAN M C等[28]分别采用Wilcoxon符号秩检验和Student's T检验判断多样性(包括α和β-多样性)和土壤养分指标在组间的差异显著性。参考赵亮等[13]采用Mantel相关性检验分析不同群落间演替相关性。参考JIAO S等[15]采用随机森林回归模型拟合所有优势菌群演替变化(自变量)与各土壤养分指标变化(响应变量)间的线性关系,模型中自变量的拟合显著性由10折交叉验证联合1 000次置换检验得出。为揭示CC和UC对ET演替的贡献性,参考JIAO S等[12,29]采用β多样性分割法分别将CC和UC产生的Bray-Curtis距离从EC中分离出来,计算分离出来的距离值在EC距离值中的占比,以此获得CC和UC分别对EC演替的贡献。
2 结果与分析
2.1 总群落及核心和特有群落多样性分析
2.1.1 高通量测序结果
经测序、质控,除去序列数<4条的ASVs,最终产生6 497个ASVs,包括649 132条有效序列,平均每个样品(34 165±12 173)条序列,样品中最高序列数为78 884条,最低序列数为19 144条。为剔除样品间测序偏差,所有样本按最小序列数19 144做抽平处理。
采用交集分析法绘制本体土、根际、根面和根内4个根系组分间ASVs upset交集图,具体见图1。
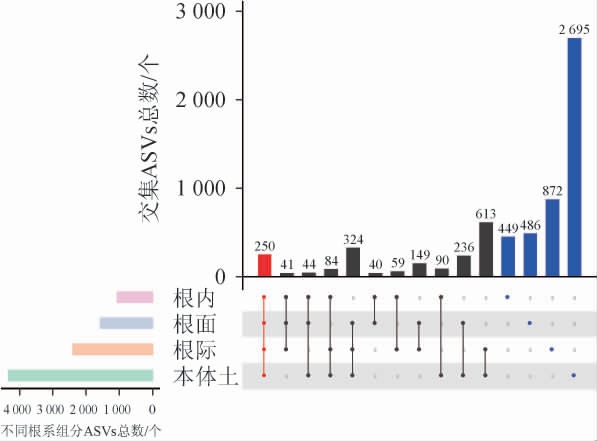
图1 本体土、根际、根面和根内ASVs upset交集图
Fig.1 Upset intersection diagram of ASVs from bulk soil,rhizosphere, rhizoplane and endosphere
由图1可知,4个根系组分共同拥有250个ASVs,ASVs总占比为3.8%,序列数总占比为55.1%。本体土、根际、根面和根内特有ASVs分别为2 695个、872个、486个和449个,共4 502个,ASVs总占比为69.3%,序列数总占比为16.4%。由此可以得出,共存于4个根系组分中的250个ASVs构成CC,且主要由高丰度的优势菌群组成;而为数众多的特有ASVs构成UC,主要由低丰度的稀有菌群组成。
2.1.2 核心群落、特有群落和总群落多样性特征
红缨子高粱根系组分中不同细菌菌群的α-多样性比较结果见图2。由图2可知,CC的Shannon指数(3.50±0.90)极显著低于EC(4.93±1.34)和UC(4.57±1.11)(P<0.01),UC(0.80±0.12)的MPD指数极显著高于EC(0.66±0.17)和CC(0.60±0.17)(P<0.01),表明UC有很高的α-多样性水平,且UC中个体间亲缘度比EC和CC中远。
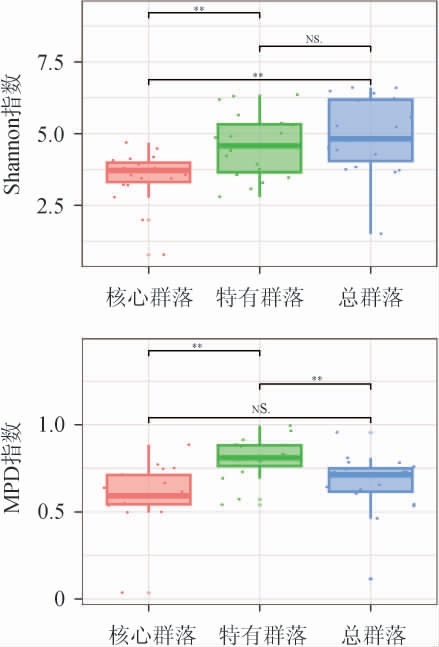
图2 红缨子高粱根系组分核心群落、特有群落和总群落之间α-多样性比较
Fig.2 Comparisons of α-diversities between core, unique and entire bacterial communities within root system of sorghum cultivar Hongyingzi
“NS.”表示差异不显著(P>0.05);“*”表示差异显著(P<0.05);“**”表示差异极显著(P<0.01)。下同。
β多样性贡献分析结果显示,CC对EC的演替贡献为49.1%,UC对EC的演替贡献为20.0%,表明CC主要决定EC演替的强度。红缨子高粱根系组分中不同细菌菌群的β-多样性比较结果见图3。由图3可知,CC的演替强度即β-多样性水平极显著高于UC(P<0.001);且Mantel相关性分析表明,CC的β多样性与EC极显著正相关(R=0.45,P=0.003),UC的β多样性也与EC极显著正相关(R=0.49,P=0.004),而CC和UC之间无显著相关性(R=-0.40,P=0.99)。说明UC和CC均能影响EC的演替强度和方向,但UC和CC之间的演替相对独立,无关联性。
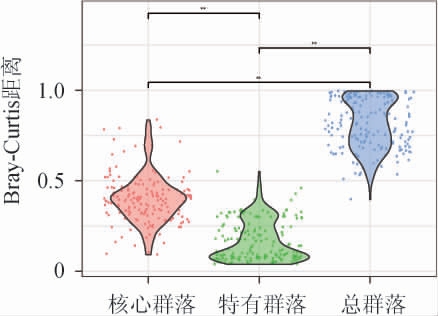
图3 红缨子高粱根系组分核心群落、特有群落和总群落之间β-多样性比较
Fig.3 Comparisons of β-diversities between core, unique and entire bacterial communities within root system of sorghum cultivar Hongyingzi
针对总群落中不同子群落的作用和角色,先前已有众多报道,其重点主要围绕稀有微生物和优势微生物进行研究。如JIAO S等[12]对原油污染土壤微生物生态地理格局进行研究,发现稀有菌群呈不平衡分布格局,组装过程机制随机化,对总群落生态稳定性发挥作用;优势菌群呈均一化分布格局,组装过程由土壤因素决定的确定性选择机制,菌群间网络关系紧密,并在总群落网络关系中处于中心地位。之后JIAO S等[11]又对农田土壤进行研究,发现优势菌群相比稀有菌群,其系统进化演替受地理距离的影响更大,且前后两种群落组装机制分别受土壤pH和年平均温度的影响。ZHAO Z Z等[10]研究带有覆盖作物的果园土壤时发现,优势菌群和稀有菌群的组装机制不同,分别是随机性和确定性机制,且它们对覆盖作物和土壤因子的响应也呈现出很大的差异。这些研究表明,总群落中的亚群落有着较为独立的多样性特征和生态角色,在它们的综合作用下,引导并维持总群落演替和其生态稳定性。
基于上述对子群落的研究基础,本研究以本体土和3种根微域为划分基础,通过交集分析将总群落划分成核心和特有两种子群落,并将它们与总群落作比较,分析它们在生态关系中的作用。结果发现,核心群落以优势群菌为主,并且与总群落演替(β-多样性)极显著相关(P<0.01),对总群落演替贡献较大(49.1%),证明其在总群落演替中的核心地位。同时也与其他领域如海草根际[30]、鲈鱼表皮[31]、草坪根际[32]和甘蔗[33]等方面的研究结论类似,核心群落由优势微生物构成,且功能和群落结构对实验处理响应与总群落相似。另外,寄居于本体土和3种根微域的特有群落,其特征与核心群落相反,种类(ASVs)数量占优势(69.3%),丰度占比(16.4%)较低,主要由稀有菌群构成。据文献报道,目前针对特有菌群的研究鲜有报道,而本研究较为优先的确定了特有群落组成结构,同时发现特有群落对总群落演替贡献相对较低(20.0%),但群落成分多样性(Shannon指数)与总群落相似,系统进化多样性(MPD指数)显著高于总群落。说明特有群落虽然不是总群落演替的主要推动力,但在群落成分和谱系构成上对维持总群落α多样性水平起关键作用。
2.1.3 群落结构分析
从EC中共注释到8个优势细菌门(平均相对丰度>1%),结果见表1。由表1可知,8个优势细菌门分别为变形杆菌门(Proteobacteria)(59.4%)、拟杆菌门(Bacteroidota)(14.2%)、酸杆菌门(Acidobacteriota)(8.3%)、放线菌门(Actinobacteriota)(8.3%)、厚壁菌门(Firmicutes)(4.0%)、芽单胞菌门(Gemmatimonadota)(2.5%)、绿弯菌门(Chloroflexi)(2.2%)和粘菌门(Myxococcota)(1.3%),这些优势细菌门在不同群落内各根系组分间相对丰度存在一定差异。如在EC中,本体土和根际中Acidobacteriota相对丰度显著高于根面和根内(P<0.05),同时根面显著高于根内(P<0.05);Acidobacteriota在CC中与EC情况类似,只是根面和根内无显著差异(P>0.05);对于UC,本体土和根际中Acidobacteriota相对丰度显著高于根内(P<0.05)。EC和CC中Firmicutes的相对丰度在根系组分间均无显著差异(P>0.05),而UC中Firmicutes在根面的相对丰度显著高于本体土(P<0.05)。结果表明,CC、UC和EC在根系各组分有其特定的结构分布特征。8种优势细菌门在韭菜种植土[34]、牛肝菌子实体根际[35]、可可种植土[36]、咖啡根际和根内[37]以及温室土壤[38]等研究中常被发现,且优势度明显,表明这些菌群可能是土壤以及植物根系的主要构成菌群[9]。优势度最高的Proteobacteria和Bacteroidota,以及优势度次之的Actinobacteriota在EC、CC或UC中,其相对丰度在根系组分间无显著差异(P>0.05),说明这四类菌群可被红缨子高粱根系高度接纳,普遍寄居于以根际、根面和根内为代表的根系各生态位中。与此相比,其他各优势菌群在根系组分间均体现出一定的丰度差异性,说明根系各生态位对这些菌群的选择性更强。
表1 不同细菌群落中8种优势细菌门在根系组分之间相对丰度差异性比较
Table 1 Differential comparisons of relative abundances between root compartments for core, unique and entire bacterial communities
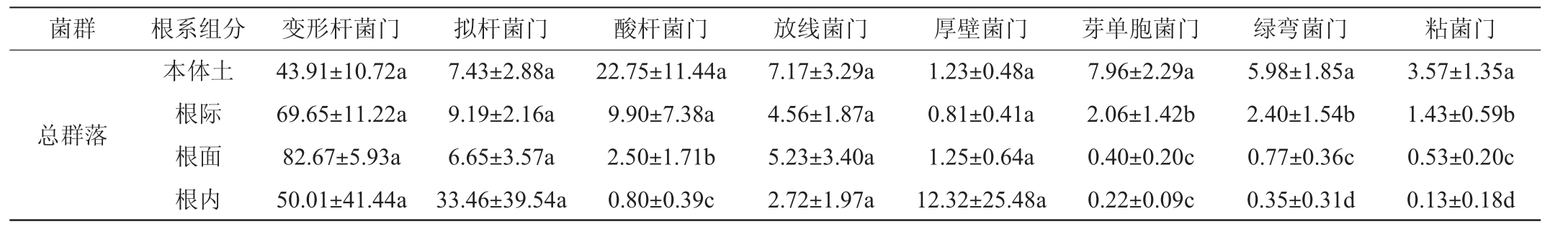
菌群 根系组分 变形杆菌门 拟杆菌门 酸杆菌门 放线菌门 厚壁菌门 芽单胞菌门 绿弯菌门 粘菌门总群落本体土根际根面根内43.91±10.72a 69.65±11.22a 82.67±5.93a 50.01±41.44a 7.43±2.88a 9.19±2.16a 6.65±3.57a 33.46±39.54a 22.75±11.44a 9.90±7.38a 2.50±1.71b 0.80±0.39c 7.17±3.29a 4.56±1.87a 5.23±3.40a 2.72±1.97a 1.23±0.48a 0.81±0.41a 1.25±0.64a 12.32±25.48a 7.96±2.29a 2.06±1.42b 0.40±0.20c 0.22±0.09c 5.98±1.85a 2.40±1.54b 0.77±0.36c 0.35±0.31d 3.57±1.35a 1.43±0.59b 0.53±0.20c 0.13±0.18d
续表

注:同一群落内,同列不同字母表示差异显著(P<0.05)。
菌群 根系组分 变形杆菌门 拟杆菌门 酸杆菌门 放线菌门 厚壁菌门 芽单胞菌门 绿弯菌门 粘菌门核心群落特有群落本体土根际根面根内本体土根际根面根内67.21±11.94a 87.12±6.66a 87.90±7.45a 54.24±40.03a 32.09±9.49a 41.71±17.12a 50.61±15.89a 40.61±32.12a 4.68±3.80a 3.05±0.62a 4.52±3.67a 42.57±41.19a 10.52±3.36a 18.32±6.97a 18.21±8.95a 34.27±30.80a 13.69±9.08a 3.74±3.01a 0.72±0.26b 0.69±0.84b 24.30±13.21a 13.05±10.56ab 8.16±8.71bc 1.68±0.70c 4.88±3.29a 3.43±2.68a 5.29±4.63a 1.32±0.87a 10.25±6.88a 9.94±2.68a 10.58±3.84a 5.00±3.29a 1.21±0.90a 0.71±0.66a 0.95±1.07a 0.93±0.84a 0.89±0.95b 1.42±0.80ab 4.59±4.03a 16.39±32.06ab 4.70±2.42a 0.73±0.48b 0.19±0.11c 0.10±0.08c 7.78±2.26a 3.12±1.47b 0.70±0.89c 0.29±0.34c 3.25±2.34a 0.91±0.69b 0.28±0.11c 0.09±0.09d 8.38±2.79a 6.89±5.26a 4.49±2.9ab 1.34±1.83b 0.37±0.32a 0.31±0.21a 0.15±0.20a 0.06±0.11a 5.78±1.95a 5.55±3.22ab 2.65±1.95bc 0.42±0.41c
2.2 细菌群落对土壤养分指标的影响
2.2.1 土壤养分指标分析
根际土和本体土间15个养分指标值的差异性见图4。由图4可知,绝大多数指标值表现为根际土高于本体土,且Mn、Zn、Cu、noN、nhN、effN和TOC在根际土中的含量显著高于本体土(P<0.05),表明根际效应显著。
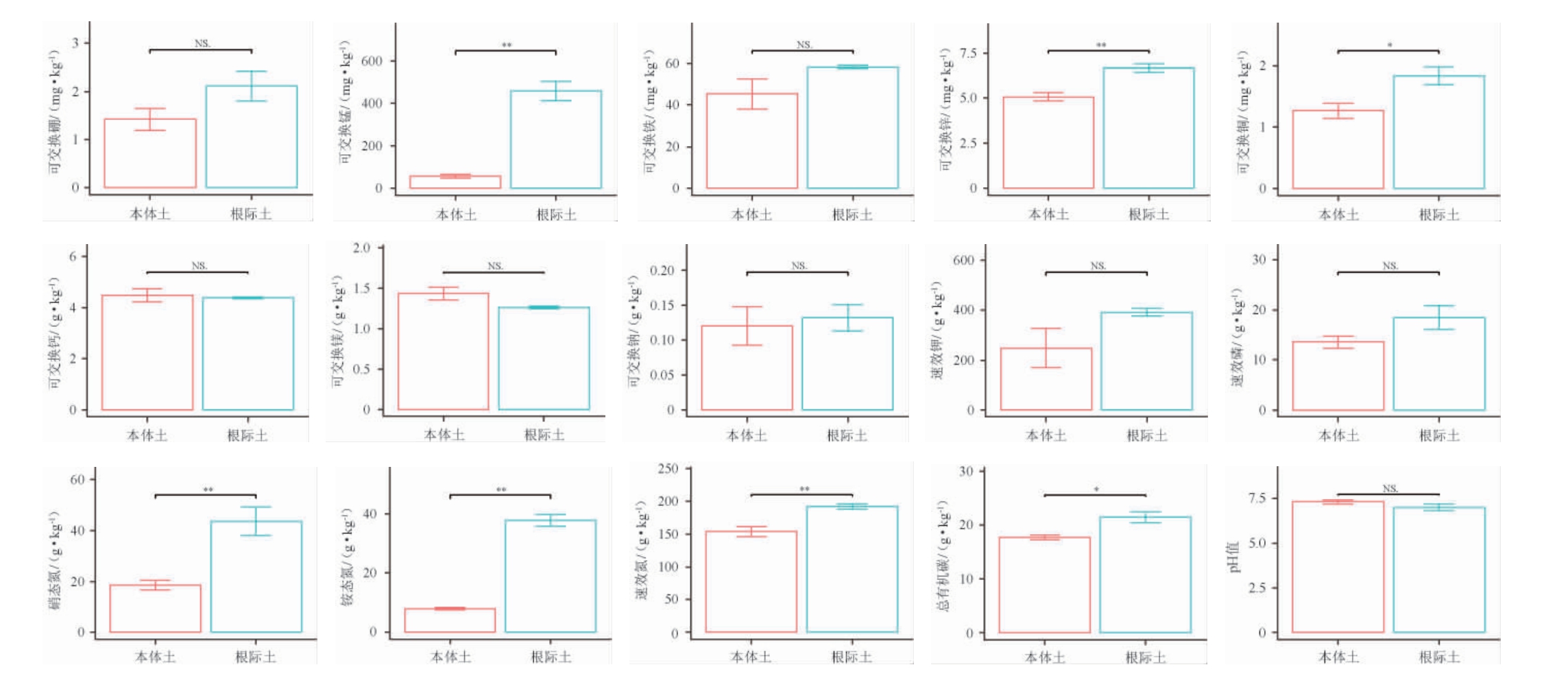
图4 红缨子高粱本体土和根际土间土壤养分指标差异性比较
Fig.4 Difference comparisons of nutrients indices between bulk soil and rhizosphere soil within root system of sorghum cultivar Hongyingzi
2.2.2 菌群演替对土壤养分的影响
为进一步探索不同细菌群落(EC、CC和UC)中,细菌菌群从土壤分别向根际、根面和根内演替迁移对根系土壤养分的影响,将本体土菌群相对丰度数据分别和根际、根面和根内菌群相对丰度数据组成数据集,然后将本体土和根际土样品的养分指标值,以及各数据集中8种优势细菌门的相对丰度逐个转化成欧式距离,以此分别作为土壤养分指标差异度和菌群丰度差异度。以8个菌群丰度差异度为自变量,以每个土壤养分指标差异度为因变量,构建随机森林回归模型,具有显著性的自变量见图5。
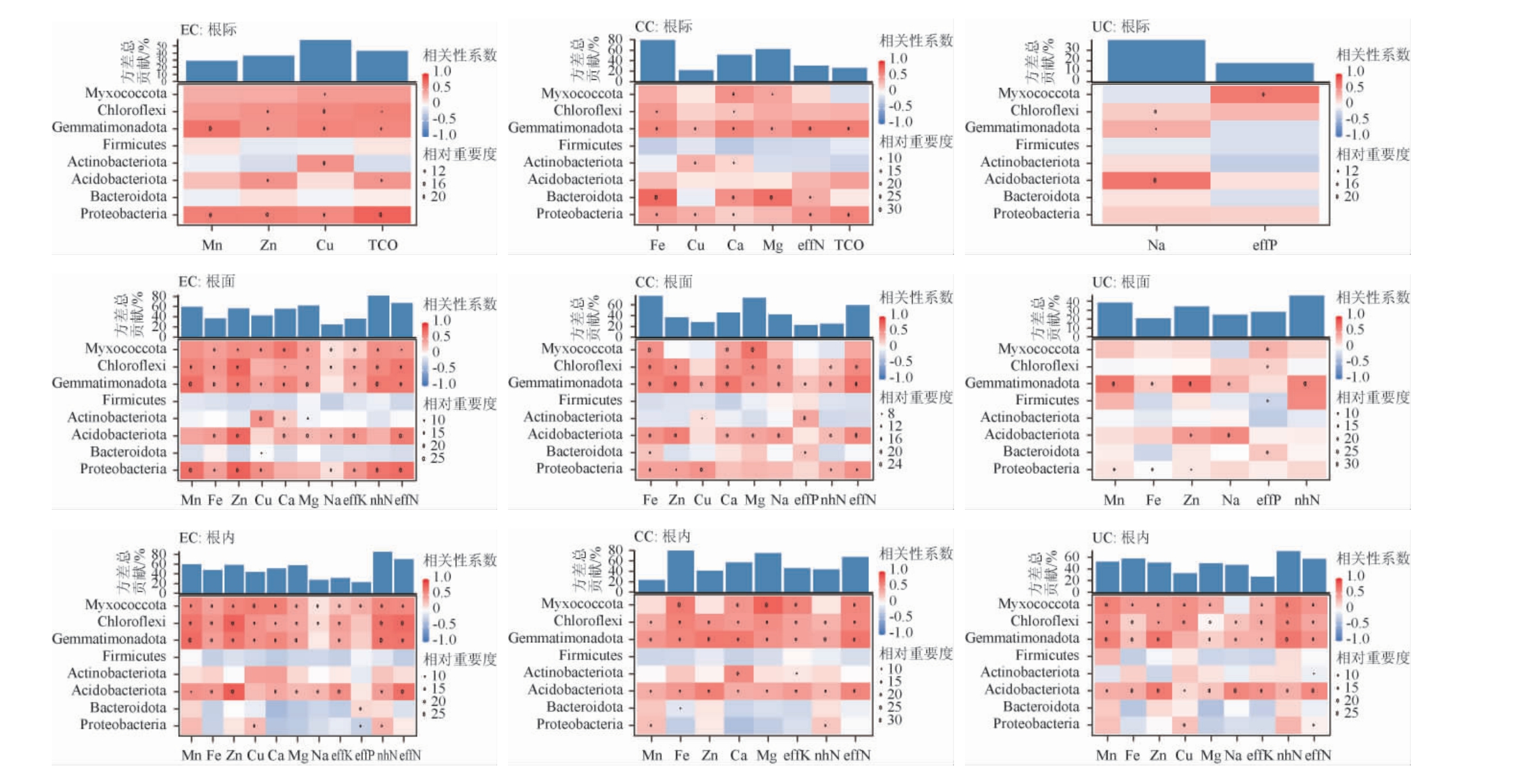
图5 不同细菌群落中8种优势细菌门与土壤养分指标间随机森林回归分析结果
Fig.5 Results of random forest regression analysis between 8 dominant phyla in different bacterial communities and soil nutrient indices
由图5可知,细菌菌群越向根组织迁移,对土壤养分影响程度越高。如UC中菌群分别向根际、根面和根内迁移,可显著影响的养分指标分别有2种(方差总贡献17.66%~39.38%)、6种(20.96%~46.43%)和9种(26.4%~70.11%),且方差总共献依次增多(P<0.05);EC和CC也表现出相同的趋势。Acidobacteriota、Gemmatimonadota、Chloroflexi和Myxococcota这4类菌群虽然相对丰度在8个菌群中优势度不高,但在EC、CC和UC中向任何根系部位迁移,均对土壤养分指标存在不同程度的影响,说明它们中的多数菌种对土壤养分调节起着关键作用。Proteobacteria在EC、CC和UC中相对丰度最高,在EC和CC中向根际和根面的迁移,会对土壤养分变化产生较大影响,说明这类菌群主要在根组织外侧调节根系土壤养分变化。与氮、磷、钾循环有关的TOC、effN、noN、nhN、effP和effK主要受向根面和根内迁移菌群的影响,而与碳循环有关的TOC仅受EC和CC中向根际迁移菌群的影响,表明向根际迁移的菌群可能主要与碳元素循环有关,而向根面和根内迁移的菌群可能与氮、磷、钾元素循环有关。在可交换离子中,Mn、Zn、Cu、Ca、Mg和Fe可作为被调节的主要离子,受3种群落不同程度的影响。
综上,从土壤-根际-根面-根内空间方向上,越向根内迁移的菌群,对土壤养分指标的影响越明显。先前针对根系组分的研究发现[6,9,39-41],微生物多样性从土壤向根内的空间方向上呈梯度下降趋势,这种趋势主要来自于植物体选择性“招募”,而根面是重要的选择屏障。据此,可认为根系生态位越接近根组织,其中寄居的微生物对植物体产生的作用越显著,进而对根系土壤养分的调节作用越明显。此外,还发现与氮、磷、钾循环有关的养分指标,主要受向根内和根面迁移菌群的影响,而与碳循环有关的指标,主要受向根际迁移菌群的影响。说明从土壤向根际、根面和根内迁移的菌群,对根系土壤养分行使着不同的调节功能。据此,可为将来开发植物促生菌提供一定的筛选参考。
3 结论
红缨子高粱根系组分中的核心群落以优势菌群为主、特有群落以稀有菌群为主。核心群落和特有群落均可带动总群落演替,其中核心群落是总群落演替的主要推动力,特有群落是总群落α多样性水平的重要驱动力。总群落中发现8种优势细菌门,可能是土壤和植物中普遍存在的菌群,它们在核心群落、特有群落和总群落间分布差异明显,且Acidobacteriota、Gemmatimonadota、Chloroflexi和Myxococcota四类菌群是调节根系土壤养分指标的主要菌群。同时,越向根部迁移的菌群(如根面和根内),对根系土壤养分指标,特别是与氮、磷、钾循环有关的指标,作用越明显;而向根际迁移的菌群(距离根较远),对根系土壤碳循环影响显著。本研究结果可为进一步开发红缨子高粱促生菌提供筛选依据。
[1]卢庆善,邹剑秋,朱凯,等.试论我国高梁产业发展——一论全国高梁生产优势区[J].园艺与种苗,2009,29(2):78-80.
[2]杨立铮,卫迦,朱赫字.茅台酒酿酒高梁的地质环境研究[J].地学前缘,1996(2):224-233.
[3]刘婷,刘满意.茅台优质酿酒高梁根际土壤细菌的多样性[J].贵州农业科学,2018,46(6):103-106.
[4]TORSVIK V.Prokaryotic diversity-magnitude,dynamics,and controlling factors[J].Science,2002,296:1064-1066.
[5]YUN K Y,PAUL G D,CHANYARAT P L,et al.Evolutionary conservation of a core root microbiome across plant phyla along a tropical soil chronosequence[J].Nat Commun,2017,8:215.
[6]EDWARDS J, JOHNSON C, SANTOS-MEDELLÍN C, et al.Structure,variation, and assembly of the root-associated microbiomes of rice[J].Plant Biology,2015,112(8):E911-E920.
[7]WILLIAM A W, ZHAO J, NICHOLAS Y, et al.Large-scale replicated field study of maize rhizosphere identifies heritable microbes[J].Plant Biology,2018,115(28):7368-7373.
[8]XU L,NAYLORA D,DONG Z B,et al.Drought delays development of the sorghum root microbiome and enriches for monoderm bacteria[J].Plant Biology,2018,115(18):E4285-E4293.
[9]ZHAO L,LU J J,JIANG L,et al.Assembly,diversity and coexistence of bacteria communities in various rhizocompartment niches in sorghum cultivars[J].Rhizosphere,2023,27:100779.
[10]ZHAO Z Z,MA Y M,FENG T Y,et al.Assembly processes of abundant and rare microbial communities in orchard soil under a cover crop at different periods[J].Geoderma,2022,406:115543.
[11]JIAO S,LU Y H.Soil pH and temperature regulate assembly processes of abundant and rare bacterial communities in agricultural ecosystems[J].Environ Microbiol,2019,22(3):1052-1065.
[12]JIAO S, CHEN W, WEI G H.Biogeography and ecological diversity patterns of rare and abundant bacteria in oil-contaminated soils[J].Mol Ecol,2017,26(19):5305-5317.
[13]赵亮,闵卓,吴福勇,等.酱香型酒醅堆积发酵过程中优势菌群和稀有菌群对细菌总群落演替模式的影响[J].食品科学,2022,43(2):168-175.
[14]XIONG C,ZHU Y G,WANG J T,et al.Host selection shapes crop microbiome assembly and network complexity[J].New Phytol,2021,229(2):1091-1104.
[15]JIAO S,YANG Y,XU Y,et al.Balance between community assembly processes mediates species coexistence in agricultural soil microbiomes across eastern China[J].ISME,2020,14:202-216.
[16]BAO S D.Soil and Agricultural Chemistry Analysis[M].Beijing:Agriculture Publication,2000:355-356.
[17]FADROSH D W, MA B, GAJER P, et al.An improved dual-indexing approach for multiplexed 16S rRNA gene sequencing on the Illumina MiSeq platform[J].Microbiome,2014,2(1):6.
[18]CHEN S F,ZHOU Y Q,CHEN Y R,et al.fastp:an ultra-fast all-in-one FASTQ preprocessor[J].Bioinformatics,2018,34(17):i884-i890.
[19]BOLYEN E,RIDEOUT J R,DILLON M R,et al.Reproducible,interactive,scalable and extensible microbiome data science using QIIME 2[J].Nat Biotechnol,2019,37(8):852-857.
[20]CALLAHAN B J,MCMURDIE P J,ROSEN M J,et al.DADA2:highresolution sample inference from Illumina amplicon data[J].Nat Methods,2016,13(7):581-583.
[21]STACKEBRANDT E, GOODFELLOW M.Nucleic acid techniques in bacterial systematics[M].New York:John Wiley and Sons,1991:115-175.
[22]BOKULICH N A, KAEHLER B D, RIDEOUT J R, et al.Optimizing taxonomic classification of marker-gene amplicon sequences with QIIME 2's q2-feature-classifier plugin[J].Microbiome,2018,6(1):90.
[23]KATOH K, STANDLEY D M.MAFFT multiple sequence alignment software version 7:improvements in performance and usability[J].Mol Biol Evol,2013,30:772-780.
[24]PRICE M N,DEHAL P S,ARKIN A P,et al.FastTree 2-approximately maximum-likelihood trees for large alignments[J].PLoS One,2010,5(3):e9490.
[25]R Core Team.R:A Language and Environment for Statistical Computing.R Foundation for Statistical Computing, Vienna, Austria, 2023, https://www.R-project.org.
[26]赵亮,王新叶,罗贞标,等.制曲原料中添加绿茶对大曲发酵过程理化特征及细菌多样性的影响[J].食品科学,2024,45(5):59-68.
[27]WEBB C,ACKERLY D,MCPEEK M,et al.Phylogenies and community ecology[J].Annu Rev Ecol Syst,2002,33:475-505.
[28]FAN M C,LIN Y B,HUO H B,et al.Microbial communities in riparian soils of a settling pond for mine drainage treatment[J].Water Res,2016,96:198-207.
[29]SHADE A,JONES S E,CAPORASO J G,et al.Conditionally rare taxa disproportionately contribute to temporal changes in microbial diversity[J].mBio,2014,5:e01371-01314.
[30]LING J,ZHOU W G,YANG Q S,et al.Spatial and species variations of bacterial community structure and putative function in seagrass rhizosphere sediment[J].Life-Basel,2021,11(8):852.
[31]DANIELA R,MARCOS P L,RICARDO S,et al.Monitoring infection and antibiotic treatment in the skin microbiota of farmed European seabass(Dicentrarchus Labrax)fingerlings[J].Microb Ecol,2022,83(3):789-797.
[32]FU J J,LUO Y L,SUN P Y,et al.Effects of shade stress on turfgrasses morphophysiology and rhizosphere soil bacterial communities[J].BMC Plant Biol,2020,20:92.
[33]RAFAEL S C S, VAANER K O, JADERSON S L A, et al.Unlocking the bacterial and fungal communities assemblages of sugarcane[J].Sci Rep,2016,6:28774.
[34]GU Y,WANG Y,WANG P Z,et al.Study on the diversity of fungal and bacterial communities in continuous cropping fields of Chinese chives(Allium tuberosum)[J].Biomed Res Int,2020,2020:3589758.
[35]ZHOU Y,SHI Z C,PANG Q L,et al.Responses of bacterial community structure, diversity, and chemical properties in the rhizosphere soil on fruiting-body formation of Suillus luteus[J].Microorganisms,2022,10(10):2059.
[36]PEDRO F F C,LUCAS P V,HOWARD J,et al.Theobroma cacao L.agricultural soils with natural low and high cadmium (Cd) in Santander(Colombia),contain a persistent shared bacterial composition shaped by multiple soil variables and bacterial isolates highly resistant to Cd concentrations[J].Curr Res Microb Sci,2021,2:100086.
[37]DINH M T.Taxonomic and functional profiles of Coffea canephora endophytic microbiome in the Central Highlands region,Vietnam,revealed by analysis of 16S rRNA metagenomics sequence data[J].Data Brief,2022,43:108372.
[38]ZHANG L L,NIU J F,LU X W,et al.Dosage effects of organic manure on bacterial community assemblage and phosphorus transformation profiles in greenhouse soil[J].Front Microbiol,2023,14:1188167.
[39]BAI L F,ZHANG X Q,LI B Z,et al.Fungal communities are more sensitive to nitrogen fertilization than bacteria in different spatial structures of silage maize under short-term nitrogen fertilization[J].Appl Soil Ecol,2022,170:104275.
[40]LI B Z,ZHANG Q Q,CHEN Y H,et al.Different crop rotation systems change the rhizosphere bacterial community structure of Astragalus membranaceus(Fisch)Bge.var.mongholicus(Bge.)Hsiao[J].Appl Soil Ecol,2021,166:104003.
[41]XIAO X,CHEN W M,ZONG L,et al.Two cultivated legume plants reveal the enrichment process of the microbiome in the rhizocompartments[J].Mol Ecol,2017,26:1641-1651.